Transmission de deux gènes liés : Estimation de la distance entre les gènes
La distance génétique est la distance qui sépare deux gènes sur un même chromosome. Sturtevant1, un élève de Morgan, a exprimé la distance entre les gènes en unités cartographiques. De nos jours, on emploie plutôt le terme Centimorgan(cM) en l'honneur de Morgan.
Calcul de la distance génétique
La distance génétique se calcule à partir de la fréquence de recombinaison ( ) , égale à :
) , égale à :

avec  = nombre de produits recombinés et
= nombre de produits recombinés et 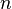 = nombre total de produits
= nombre total de produits
L'unité de distance génétique est le (Cm) comme vu chapitre(Définition). Elle correspond à un segment de chromatide sur lequel la probabilité qu'un crossing-over s'effectue est de 1 %. Le morgan est donc l'intervalle où se produit en moyenne un événement de recombinaison par méiose. La distance génétique augmente avec la taille de l'intervalle séparant deux marqueurs : lorsque la distance physique grandit, la probabilité qu'il se produise un crossing-over grandit également. Néanmoins, la relation entre la fréquence des remaniements consécutifs aux crossing-over et la fréquence de recombinaison n'est pas toujours linéaire. Elle dépend de la position relative des marqueurs en jeu. On peut arbitrairement distinguer trois situations:
1. Les deux marqueurs sont proches : sur le segment chromosomique compris entre ces deux marqueurs, il ne se produit jamais plus qu'un crossing-over par méiose; la fréquence de recombinaison reflète alors directement la fréquence des remaniements consécutifs aux crossing-over. Tous les produits de la méiose qui ont une hérité une chromatide qui a été touchée par un crossing-over(et seulement ceux-là) sont recombinés.Donc dans ce cas, la distance génétique est égale au pourcentage de produits recombinés.
2. Les deux marqueurs sont plus éloignés que dans le cas précédent : sur le segment chromosomique compris entre ces deux marqueurs, il peut y avoir soit aucun, soit un seul, soit plusieurs crossing-over par méiose. La fréquence de recombinaisons sous-estime alors la fréquence de remaniements consécutifs aux crossing-over multiples. En effet, lorsque ces derniers affectent les deux chromatides, ils restaurent des associations parentales et ne peuvent donc pas être décelés; quand ils touchent trois des quatre chromatides, seulement deux chromatides donnent des produits recombinés, comme si seulement un crossing-over s'était produit. Donc dans ce cas, il est nécessaire pour calculer correctement la distance génétique entre les deux marqueurs d'utiliser des méthodes permettant d'estimer la fréquence des crossing-over multiples.
3. Les deux marqueurs sont situés à une distance telle qu'il se produit toujours au moins un crossing-over entre eux. À la méiose, les simples crossing-over produisent deux produits parentaux et deux produits recombinés. De même, les crossing-over multiples, affectant de manière aléatoire les quatre chromatides, produisent globalement autant de produits parentaux que de produits recombinés. En conséquence, la fréquence totale des produits recombinés est égale à 0.5. Donc dans ce cas, les deux marqueurs sont généralement indépendants. Il est impossible de calculer une distance génétique.
En résumé, les distances génétiques ne peuvent être calculées de manière fiable que pour les fréquences de recombinaison faibles, dans des intervalles assez petits pour que les crossing-over multiples soient absents. Si la fréquence de produits recombinés est supérieure à une certaine valeur(de l'ordre de 0,1), il faut alors rechercher des marqueurs intermédiaires. Si on ne dispose pas de tels marqueurs, il faut utiliser des méthodes de calcul de distances génétiques permettant d'estimer la fraction de crossing-over multiples.
Application
La distance génétique du génome (mesurée en centimorgans) consiste donc à localiser sur les chromosomes la région en cause dans une maladie génétique. Une telle distance génétique a permis de placer et organiser une multitude de marqueurs polymorphes sur les différents chromosomes.
Ces marqueurs polymorphes, utilisés dans les études génétiques sont :
- des micro- ou minisatellites
- des SNP(single nucleotide polymorphism), qui, comme leur nom l'indique :
- concernent un seul nucléotide en une position donnée du génome,
- dont la nature est variable entre les individus d'une même espèce,
- sans pourtant avoir la moindre conséquence sur l'expression de gènes.
Aujourd'hui, ces différentes distances sont unifiées pour plusieurs organismes et l'on peut voir dans les banques de données la localisation des gènes, marqueurs et maladies sur les différents chromosomes d'une espèce.
